L’objectif de cette évaluation était de déterminer et comparer dans deux études parallèles les performances diagnostiques de :
- neuf trousses commerciales de détection des norovirus (NoV) dans les selles par technique de RT-PCR en temps réel.
- huit trousses commerciales de détection des rotavirus du groupe A (RVA) dans les selles par technique de RT-PCR en temps réel.
Remarque : quatre trousses étaient des trousses multiplexes, qui étaient donc communes aux deux évaluations.
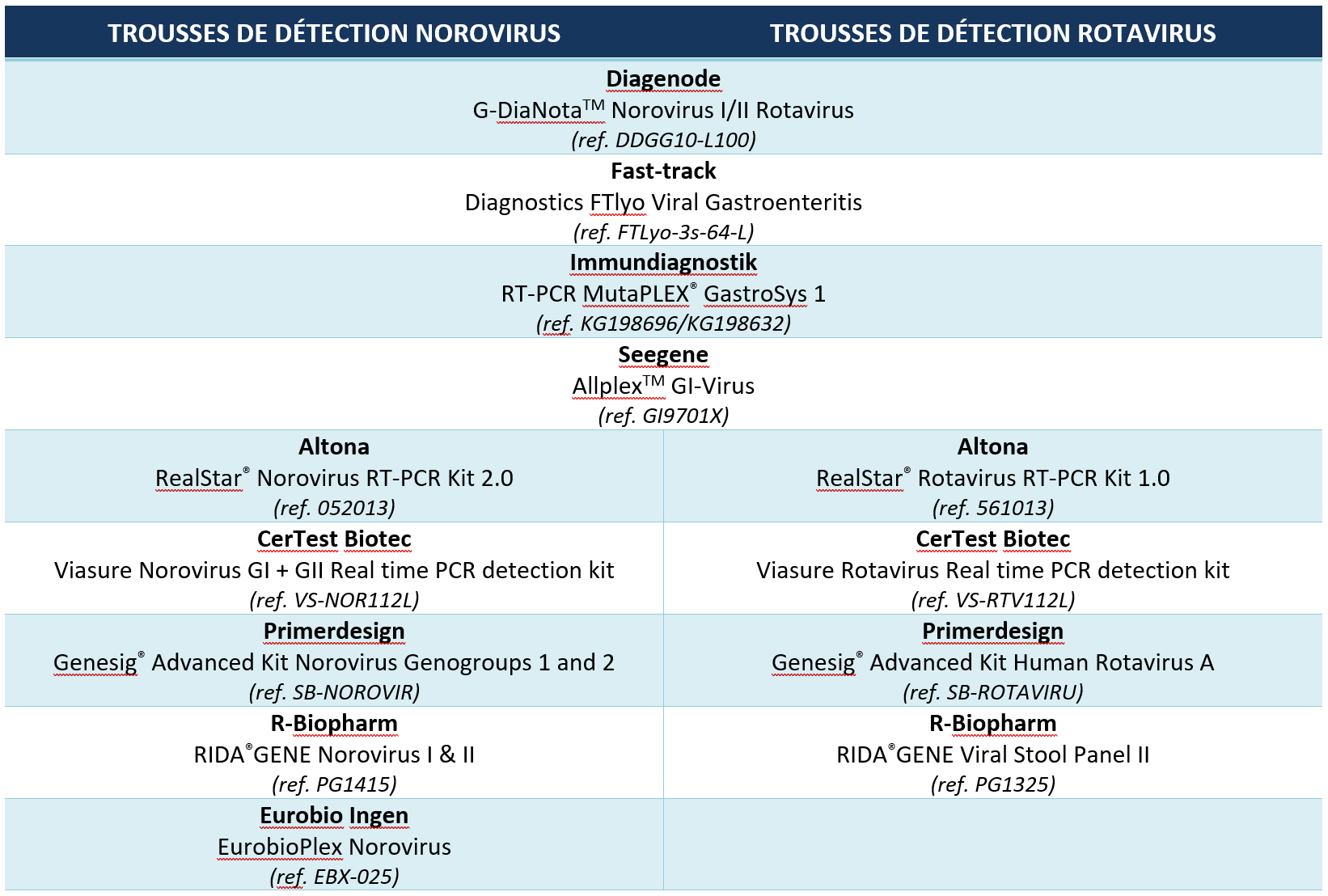
Les trousses évaluées ont été sélectionnées parmi celles disponibles sur le marché français. Seuls les fabricants ou distributeurs de trousses qui acceptaient de participer à cette étude de façon volontaire, de fournir gracieusement au CNR virus des gastro-entérites les trousses de détection nécessaires à cette évaluation et qui acceptaient que le CNR virus des gastro-entérites puissent communiquer librement les résultats de cette évaluation, ont été retenus. L'évaluation a porté sur les trousses suivantes :
Les essais ont été réalisés sur des échantillons de selles humaines issus de la collection du CNR virus des gastro-entérites (conservées à -40°C), sélectionnés afin de représenter les souches virales les plus importantes sur le plan épidémiologique et répartis comme suit :
- 50 selles positives en NoV (dont 3 co-infections avec du RVA et 1 co-infection NoV GI, NoV GII et RVA) incluses dans l’évaluation des trousses de détection des NoV : 25 souches de génogroupe I (5 GI.1, 5 GI.2, 5 GI.4, 1 GI.6, 4 GI.6[P11] et 5 GI.7) et 26 souches de génogroupe II (1 GII.3, 5 GII.4, 5 GII.6[P7], 4 GII.2[P16], 5 GII.4[P16], 5 GII.17 et 1 non typable).
- 53 selles positives en RVA (dont 4 échantillons co-infectés avec du NoV) incluses dans l’évaluation des trousses de détection des RVA : 15 souches G1P[8], 10 G2P[4], 5 G3P[8], 5 G4P[8], 11 G9P[8], 5 G12P[8] et 2 souches non typables (G-UDP-UD).
- 49 selles négatives en NoV et RVA, incluses en parallèle dans les deux évaluations.
Au cours de l’évaluation, la présence ou l'absence d’ARN de norovirus et de rotavirus du groupe A a été vérifiée par les méthodes de référence utilisées en routine au CNR (RT-PCR en temps réel adaptées de la littérature par le CNR) :
- pour tous les échantillons positifs (norovirus et /ou rotavirus) de la collection du CNR,
- pour tout échantillon négatif de la collection du CNR, lorsque au moins l’un des tests de RT-PCR évalué donnait un résultat positif.
La sensibilité, la spécificité, les rapports de vraisemblance positif et négatif (LR+ et LR-) ainsi que le diagnostic odds ratio (DOR) ont été calculés avec leurs intervalles de confiance à 95% (IC 95%) en utilisant le logiciel Meta-DiSc (Zamora et al, BMC Medical Research Methodology 2006).
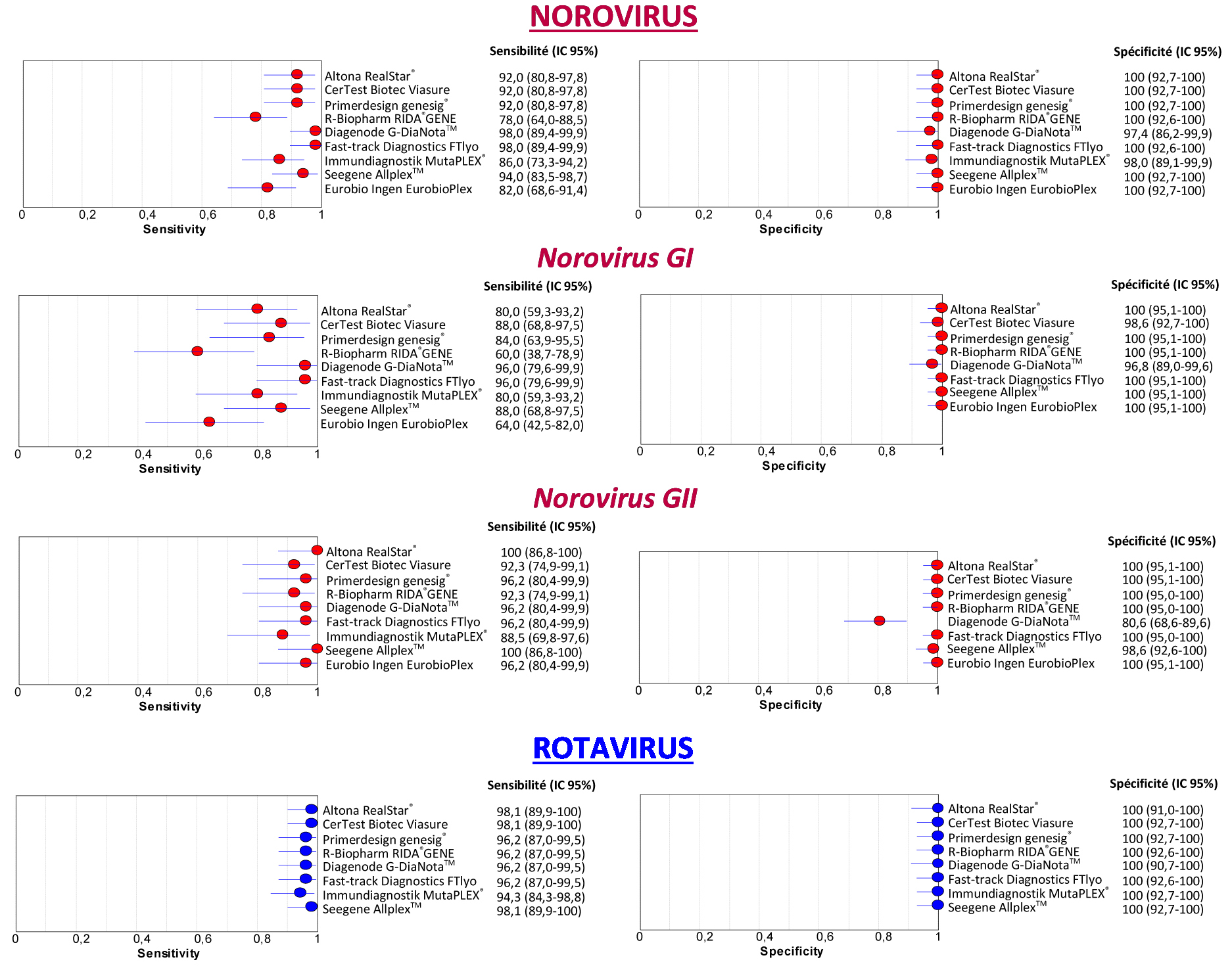
Concernant la détection des RVA, les huit trousses évaluées présentaient une sensibilité élevée (94,3-98,1%) et une excellente spécificité (100%) (Figure 1). Des LR+ élevés (74,4-97,2), des LR- faibles (0.028-0.065) et des DOR très élevés (1428,4-3465) étaient observés (Tableau 1). Ces données indiquaient une association forte entre les résultats des tests RT-PCR évalués et la présence de rotavirus, les tests évalués étaient donc hautement discriminants et adaptés au diagnostic des infections à rotavirus. Les IC 95% de l’ensemble des indicateurs de performance diagnostique se chevauchaient, la performance diagnostique pour la détection des rotavirus était donc similaire pour les huit trousses évaluées.
Concernant la détection des NoV, les neuf trousses évaluées présentaient une sensibilité moyenne (78,0% pour le kit R-Biopharm, 82,0% pour le kit Eurobio Ingen et 86,0% pour le kit Immundiagnostik), bonne (92,0% pour les kits Altona, CerTest Biotec, PrimerDesign et 94,0% pour le kit Seegene) voire excellente (98,0% pour les kits Diagenode et Fast-Track Diagnostics) (Figure 1). Ces différences de sensibilité n’étaient toutefois pas statistiquement significatives, sauf entre le kit R-Biopharm (78,0% ; IC 95% : 64,0-88,5) vs. les kits Diagenode et Fast-Track Diagnostics (98,0% ; IC 95% : 89,4-99,9). Les spécificités des neuf trousses étaient excellentes, comprises entre 97,4 et 100 % (Figure 1). Des LR+ élevés (37,2-95,1), des LR- faibles (0.021-0.228) et des DOR élevés (294,9-3201) étaient observés (Tableau 1).
Il apparait que ces performances diagnostiques étaient génogroupe-dépendantes :
- pour le génogroupe I, les sensibilités des trousses R-Biopharm et Eurobio Ingen étaient médiocres (60,0 et 64,0% respectivement) et nettement moins élevées que celles des autres trousses (80,0%-88,0%) et notamment des trousses Diagenode et Fast-Track Diagnostics qui présentaient la meilleure sensibilité (96,0%). Cependant, seule la différence de sensibilité entre le kit R-Biopharm (60,0% ; IC 95% : 38,7-78,9) et les kits Diagenode et Fast-Track Diagnostics (96,0% ; IC 95% 79,6-99,9) était statistiquement significative. Les spécificités étaient quant à elles toutes excellentes (entre 96,8 et 100%).
- concernant le génogroupe II, les sensibilités des trousses étaient élevées et similaires, comprises entre 88,5 pour Immundiagnostik et 100% pour Altona et Seegene. Les trousses présentaient toutes d’excellentes spécificités comprises entre 98,6 et 100%, sauf la trousse Diagenode qui présentait une spécificité médiocre (80,6%), statistiquement différente des autres trousses (pas de chevauchement des IC 95%). De fait, le LR+ de la trousse Diagenode était plus faible (5) que celui des autres trousses (48,4-145,3).
En conclusion, les huit trousses de détection des RVA sont toutes bien adaptées au diagnostic de ces virus dans les selles.
Les trousses Altona, CerTest Biotec, PrimerDesign, Fast-Track Diagnostics, Immundiagnostik, et Seegene sont adaptées au diagnostic des norovirus dans les selles. La sensibilité pour la détection des norovirus génogroupe I de la trousse Eurobio Ingen et surtout de la trousse R-Biopharm, ainsi que la spécificité vis-à-vis des norovirus du génogroupe II de la trousse Diagenode doivent être améliorées avant que ces trousses puissent être utilisées pour le diagnostic routinier des norovirus.
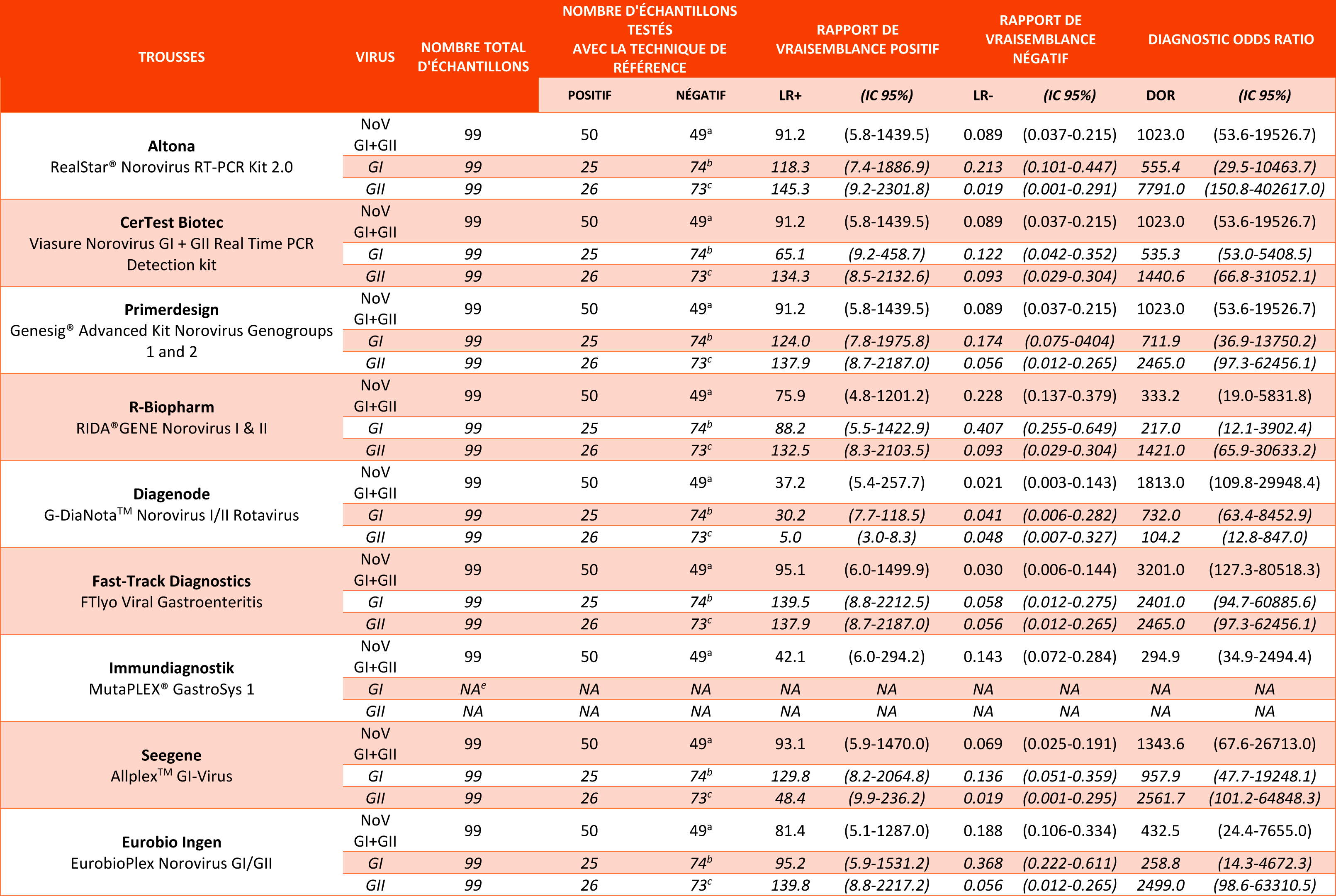
a. Les échantillons négatifs en norovirus par la technique de référence et présentant un résultat ininterprétable avec la trousse testée, du fait de la présence d'inhibiteurs de la RT-qPCR, sont exclus de l'analyse pour R-Biopharm (n=1), Diagenode (n=11) et Fast-track Diagnostics (n=1).
b. 49 échantillons norovirus génogroupe I négatifs et 25 échantillons norovirus génogroupe II positifs inclus. Les échantillons négatifs en norovirus génogroupe I par la technique de référence et présentant un résultat ininterprétable avec la trousse testée, du fait de la présence d'inhibiteurs de la RT-qPCR, sont exclus de l'analyse pour R-Biopharm (n=1), Diagenode (n=11) et Fast-track Diagnostics (n=1).
c. 49 échantillons norovirus génogroupe II négatifs et 24 échantillons norovirus génogroupe I positifs inclus. Les échantillons négatifs en norovirus génogroupe II par la technique de référence et présentant un résultat ininterprétable avec la trousse testée, du fait de la présence d'inhibiteurs de la RT-qPCR, sont exclus de l'analyse pour Primerdesign (n=1), R-Biopharm (n=1), Diagenode (n=11) et Fast-track Diagnostics (n=1).
NA : Nnn applicable (pas de différenciation des norovirus génogroupe I et des norovirus génogroupe II).
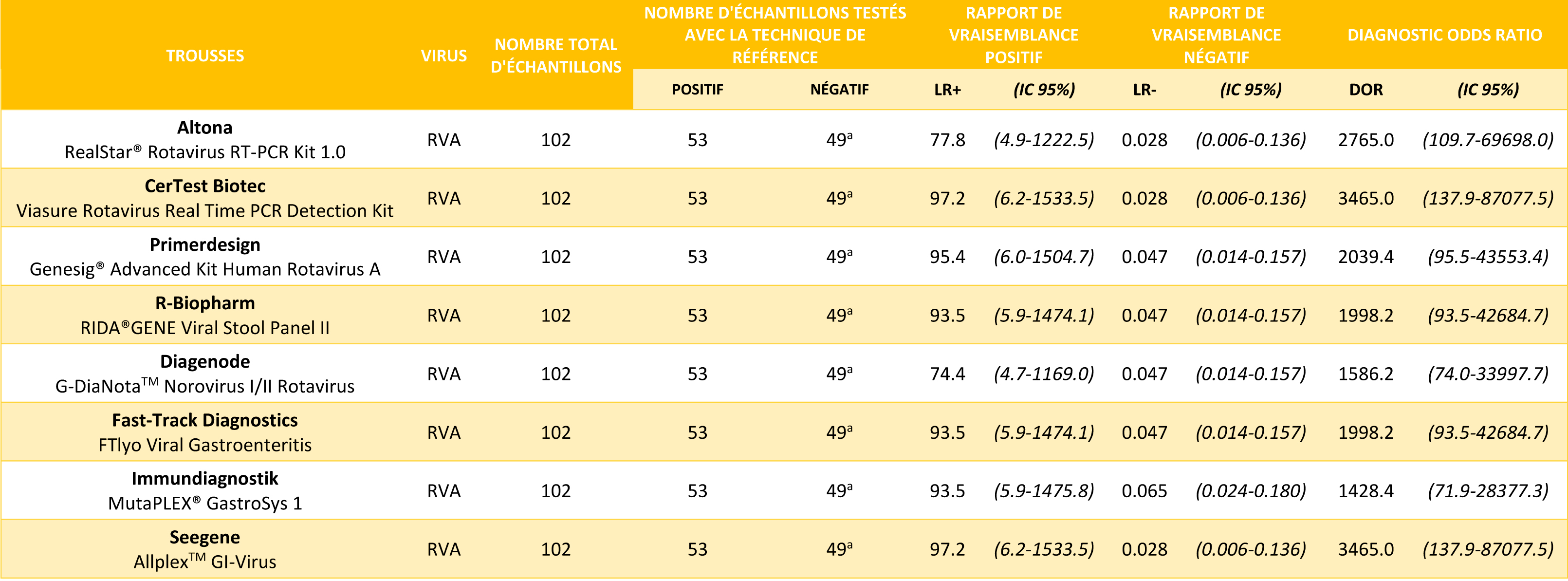
a. Les échantillons négatifs en rotavirus par la technique de référence et présentant un résultat ininterprétable avec la trousse testée, du fait de la présence d'inhibiteurs de la RT-qPCR, sont exclus de l'analyse pour Altona (n=10), R-Biopharm (n=1), Diagenode (n=11) et Fast-track Diagnostics (n=1).